Ralstonia solanacearum (strain GMI1000) (Pseudomonas solanacearum)
Taxonomy: cellular organisms; Bacteria; Proteobacteria; Betaproteobacteria; Burkholderiales; Burkholderiaceae; Ralstonia; Ralstonia solanacearum
Average proteome isoelectric point is 7.2
Get precalculated fractions of proteins
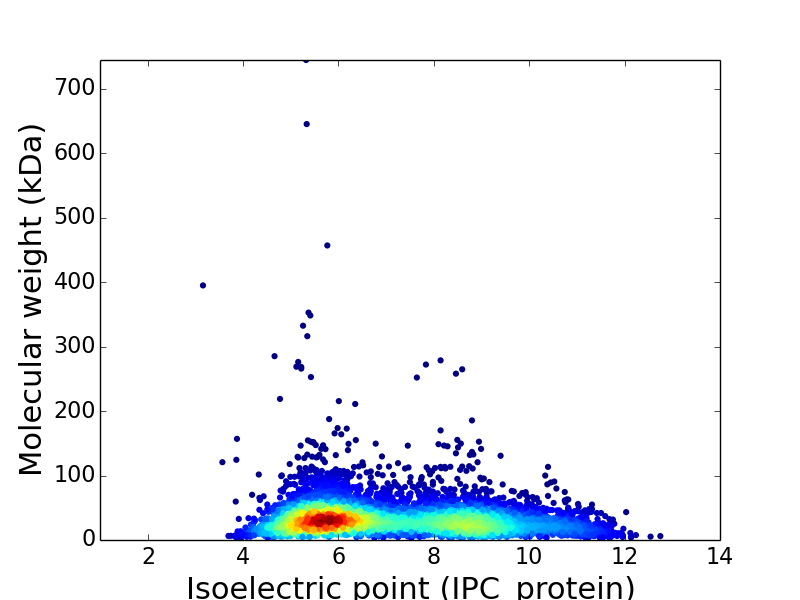
Virtual 2D-PAGE plot for 5,002 proteins (isoelectric point calculated using IPC_method)
Get csv file with sequences according given criteria:
* You can choose from 18 different methods for calculating isoelectric point
Protein with the lowest isoelectric point:
>tr|Q8XQP2|Q8XQP2_RALSO Probable hemagglutinin/hemolysin-related protein
MVSPSVLSNHTDIPVTSTTTFVTAPNGPANFLVAAHKDDVANYARSGNDLVVEFKDGHALRLQGFFANGTDFHNLVFQQEDGRMLAVFDGALTAEGDGIVDSAVTLEAIAESSSTATLLGILGAVGLGAGIAAAGGGGGGGSTAAVGAGTGSGSVAPPASVSVDSVDGRTAGSGVVTTNASPAVTGSGAV
PNAQVTVSVDGQSTGTVTADANGNWSYALTSLSDGSHQVTAIQTDSSGTSSAPTAVAFTVDTVAPASPVVASVTDDVDPVTGAIVSGSSTNDATPTLAGTAEAGSTITVYDNGTAIGTATADASGNWTFTPATALADGSHSITVTATDAAGNVSPATSAITLTVDTAAPTAPTLNPSDGTTLSGTAEAGS
TVSLDLNGDGTPDATVTADASGNWTYTPTTPLADGTVVSATATDPAGNVSPAATVTVAPTGTTLAAPVISTVTDDVAPVTGAITAGSSTNDATPTLTGTAEASSTVSVFDGTTLLGTTTADASGNWTFTPTTALTDGSHSLTATATDTAGNVSTASSAFALTVDTAAPATPVVASVTDDVAPVTGALTSG
GSTNDATPTLAGTADPNSTISIFDGTTLLGTTTADASGNWTFTPTTALTDGSHSLTATATDPAGNVSTATSAFTLTVDTAAPATPVVASVTDNVAPVTGTLTSGGSTNDTTPTLAGTAEASSTVSVFDGTTLLGTTTADASGNWTFTPTTALTDGSHSLTATATDTAGNVSTASSAFALTVDTAAPATPV
VASVTDDVAPVTGALTSGGSTNDATPTLAGTADPNSTISIFDGTTLLGTTTADASGNWTFTPTTALTDGSHSLSAAATDAAGNVGAASSAFTLTIDTAAPAIPVISTVTDNVAPVTGDITAGGSTNDTTPTLAGTAEANSTVSIFDGTTLLGTTTADASGNWTFTPTTALADGSHSLTATATDATGSVSP
ATTAFTLTVDTAAPATPVISSVTDDVAPVTGAITSGSSTNDATPTLAGTAEANSTVSIFDGTTLLGTTTSDASGSWTFTPTAALTDGSHSLTATANDAAGNVSTASSAFTLTVDTATPATPVISTVTDNVAPVTGAITAGSSTNDPTPTLAGTAEANSTVSVFDGTTLLGTTTADASGNWTFTPTTALAD
GSHSLTATATDATGSVSPATTAFTLTVDTAAPATPVIGTVTDDVAPVTGAITAGSSTNDATPTLTGTAEADTTISIFDGTTLLGTTTADALGNWTFTPTTALTDGSHSLTATATDAAGNVSAATSAFILTVDTATPATPVISTVTDNVAPVTGDITAGGSTNDAMPVLTGTAEANSTISIFDGTTLLGTT
TADASGNWTYTPTTALADGSHSLTATATDAAGNVSTATSAFTLTVDTAAPATPVISAVTDAVAPVTGTLTSGGSTNDPTPTLAGTAEANSTISILDGTTLLGTTTADASGNWSFTPTTALADGNHSLTATATDAAGNVSTATSAFTLTVDVSVPLAPVLVSVTDDVSPVTGTLTSGGSTNDATPTLAGTA
EADTTVSIFDGTTLLGTTTADALGNWTFTPTTALTDGSHSLTATATDATGSVSPATAAFTLTVDTAAPATPVIGTVTDDVAPVTGAITAGSSTNDATPTLTGTAEANSTINVFDGTTLLGTTTSDASGNWTYTPTAALTDGSHSLSATATDATGSVSPATAAFTLTVDTAAPATPIIGTVTDDVAPVTGA
ITAGGSTNDATPTLTGTAEANSTVSIFDGTTLLGTTTADASGNWSYTPTTALTDGSHSLTATATDAAGNVSPATSAFTLTVDTAAPATPVISAVTDAVAPVTGTITSGGSTNDAAPTLTGTAEANSTINVFDGTTLLGTTTADALGSWTFTPTTALIDGSHSLTATATDAAGNVSTASSAFTLTVDTAAP
ATPVIGTVTDAVAPVTGTITAGGSTNDANPTLTGIAEGNSTVSIFDGTTLLGTTTADASGNWSYTPTTALTDGSHSLTATATDAAGNVSSASSAFTLTVDTAAPAAPVIGTVTDDVAPITGTVAAGGSTNDTTPTLAGTAEANSTINVFDGTTLLGTTTADASGNWTFTPSTPLTDGSHSFTATATDAAG
NVGTASSAFTLTVDTDAPTIPVISTVTDSVAPVTGAITAGGSTNDAMPVLTGTAEANSTISVFDGTTLLGTTTADASGNWTFTPTTALTDGSHSLTATATDPAGNVSTTSSAFALTVDTTAPAAPVITTVTDAVSPVTGAITAGGSTNDAAPALTGTAEAYSTISVFDGTTLLGTTTADASGNWTFTPTT
ALTDGSHSLSATATDATGNVSTASSAFTLTVDTDAPATPVISTVTDDVAPVTGAITAGGSTNDATPTLAGTAEANSTVSIFDGTTLLGTTTADASGSWTFTPTTALTDGSHSLTATVTDAAGNVSTASTAFTLTVDTAAPAAPVLNPTTGTTLSGTAEANSTISVSDGTTLLGTTTADASGTWTYTPTTP
LASGTVVSATATDPAGNVSPAATVTVTAVNTAPAAPVISTVTDDVAPITGAITAGGSTNDATPTLTGTAEADSTISVFDGTTLLGTTTADASGNWTFTPTTALTDGSHSLTATATNATGDVSPASSAFTLTVATAAPATPVITTVTDDVAPVTGAIAAAGSTNDATPTLAGTAVANSTVSVFDGTTLLGT
TTADASGNWTFTPTTALTDGSHSFTATATDAAGNVSAASSAFTLTVDTAAPATPVVGTVTDSVAPVTGAITAGGTTNDATPTLTGTAEANSTISIFDGITLVGTTTADASGNWTFSPTTALADGSHSLTATATDAAGNVSTASSAFALTVDTAAPAAPVLVSVTDNVAPLTGTIAAGGATNDATPTLTGT
AEANSTISVFDGTTLLGTTTADASGNWTFTPTTSLADGSRSLTATTTDTAGNVSTASTAFTLTVDTDAPPTPVITTVTDNVAPVTGAVTAGGSTNDATPTLTGTAEAHNMISIFDGATLIGATVADASGNWTFTPIQPLADGNHSLTATATDPAGNVGTASSTFTLTVDTAAPAAPVISTVTDNVAPVTG
AITAGGSTNDAMPVLTGTAEANSTVSILDGTTLLGTTTADASGNWTFTPTTALTDGSHSITATATDAAGNVSTASSAFAMTVDTAAPATPVITTVTDDVAPVTGAITAGGSTNDATPTLTGTAEANSTISIFDGSTLLGTVTADASGNWTYTPTTALADGSHSFTATATDAAGNVSTATSAFTLTVDTAA
PATPALVSVTDDVAPVTGVLTSGGSTNDATPTLTGTAEANSTVSILDGTTLLGTTTADASGNWTFTPTTPLADGSHSLTATATDAAGNVSTATSAFTLTVDTAAPATPVLASVTDDVAPVTGALTSGGSTNDATPTLAGTAEANSTISILDGTTLLGTTTADASGNWTFTPTTALADGSHSLTATATDAA
GNVSTATSAFTLTVDTAAPATPVLVSVTDDVAPVTGVLTSGGSTNDATPTLTGTAEANSTISVFDGTTLLGTTTADASGNWTFTPTTGLTDGSHSLTATATDAAGNVSPATSAFTQTVDTAAPATPALVSVTDDVAPVTGVLTSGGSTNDATPTLAGTAEANSTISILDGTTLLGTTTADASGSWTFTPT
TGLTDGSHSLTATATDAAGNVSTATTAFTLTVDTVAPTTTATLTTLTADTGTTGDWITGDTAPTLSGTVSAALGTGETVQVSLDGGTTWSDAVVSGTSWSWYPVAQFAAGGYTAMTRVVDAAGNAGAASSQAFTIQTTAAPVVSVQQSAGLLGIAGANLFDLIDFSQQQLFAATDYNNNIQSVVLHYGGV
ASLLSTSQYSANSALASELGLQFSVVNNNGILGIAASSTMTITATDGGTIDNLRLNEFLGSVTLSGGGLLGINASLLSSVTIQATDSTSLSSTANAGQLLNVSLLGTTQPSAIIEGTSGNNTLTGTSGNDRLYGYAGDDTLNAGAGNDLLRGGAGNDVLNGGDGNDILIGGAGNDTLTGGAGGDIFLWEV
NAADNTGGNGADTITDFTLASGPDDATADRIDVSALLVGYTADADGAAHYVNGVATIDSGDTIGQYLTATSDGAGNTVITIDRDGAGTTYSATTLVTLTNVTTNLETLLANHQILV
MVSPSVLSNHTDIPVTSTTTFVTAPNGPANFLVAAHKDDVANYARSGNDLVVEFKDGHALRLQGFFANGTDFHNLVFQQEDGRMLAVFDGALTAEGDGIVDSAVTLEAIAESSSTATLLGILGAVGLGAGIAAAGGGGGGGSTAAVGAGTGSGSVAPPASVSVDSVDGRTAGSGVVTTNASPAVTGSGAV
PNAQVTVSVDGQSTGTVTADANGNWSYALTSLSDGSHQVTAIQTDSSGTSSAPTAVAFTVDTVAPASPVVASVTDDVDPVTGAIVSGSSTNDATPTLAGTAEAGSTITVYDNGTAIGTATADASGNWTFTPATALADGSHSITVTATDAAGNVSPATSAITLTVDTAAPTAPTLNPSDGTTLSGTAEAGS
TVSLDLNGDGTPDATVTADASGNWTYTPTTPLADGTVVSATATDPAGNVSPAATVTVAPTGTTLAAPVISTVTDDVAPVTGAITAGSSTNDATPTLTGTAEASSTVSVFDGTTLLGTTTADASGNWTFTPTTALTDGSHSLTATATDTAGNVSTASSAFALTVDTAAPATPVVASVTDDVAPVTGALTSG
GSTNDATPTLAGTADPNSTISIFDGTTLLGTTTADASGNWTFTPTTALTDGSHSLTATATDPAGNVSTATSAFTLTVDTAAPATPVVASVTDNVAPVTGTLTSGGSTNDTTPTLAGTAEASSTVSVFDGTTLLGTTTADASGNWTFTPTTALTDGSHSLTATATDTAGNVSTASSAFALTVDTAAPATPV
VASVTDDVAPVTGALTSGGSTNDATPTLAGTADPNSTISIFDGTTLLGTTTADASGNWTFTPTTALTDGSHSLSAAATDAAGNVGAASSAFTLTIDTAAPAIPVISTVTDNVAPVTGDITAGGSTNDTTPTLAGTAEANSTVSIFDGTTLLGTTTADASGNWTFTPTTALADGSHSLTATATDATGSVSP
ATTAFTLTVDTAAPATPVISSVTDDVAPVTGAITSGSSTNDATPTLAGTAEANSTVSIFDGTTLLGTTTSDASGSWTFTPTAALTDGSHSLTATANDAAGNVSTASSAFTLTVDTATPATPVISTVTDNVAPVTGAITAGSSTNDPTPTLAGTAEANSTVSVFDGTTLLGTTTADASGNWTFTPTTALAD
GSHSLTATATDATGSVSPATTAFTLTVDTAAPATPVIGTVTDDVAPVTGAITAGSSTNDATPTLTGTAEADTTISIFDGTTLLGTTTADALGNWTFTPTTALTDGSHSLTATATDAAGNVSAATSAFILTVDTATPATPVISTVTDNVAPVTGDITAGGSTNDAMPVLTGTAEANSTISIFDGTTLLGTT
TADASGNWTYTPTTALADGSHSLTATATDAAGNVSTATSAFTLTVDTAAPATPVISAVTDAVAPVTGTLTSGGSTNDPTPTLAGTAEANSTISILDGTTLLGTTTADASGNWSFTPTTALADGNHSLTATATDAAGNVSTATSAFTLTVDVSVPLAPVLVSVTDDVSPVTGTLTSGGSTNDATPTLAGTA
EADTTVSIFDGTTLLGTTTADALGNWTFTPTTALTDGSHSLTATATDATGSVSPATAAFTLTVDTAAPATPVIGTVTDDVAPVTGAITAGSSTNDATPTLTGTAEANSTINVFDGTTLLGTTTSDASGNWTYTPTAALTDGSHSLSATATDATGSVSPATAAFTLTVDTAAPATPIIGTVTDDVAPVTGA
ITAGGSTNDATPTLTGTAEANSTVSIFDGTTLLGTTTADASGNWSYTPTTALTDGSHSLTATATDAAGNVSPATSAFTLTVDTAAPATPVISAVTDAVAPVTGTITSGGSTNDAAPTLTGTAEANSTINVFDGTTLLGTTTADALGSWTFTPTTALIDGSHSLTATATDAAGNVSTASSAFTLTVDTAAP
ATPVIGTVTDAVAPVTGTITAGGSTNDANPTLTGIAEGNSTVSIFDGTTLLGTTTADASGNWSYTPTTALTDGSHSLTATATDAAGNVSSASSAFTLTVDTAAPAAPVIGTVTDDVAPITGTVAAGGSTNDTTPTLAGTAEANSTINVFDGTTLLGTTTADASGNWTFTPSTPLTDGSHSFTATATDAAG
NVGTASSAFTLTVDTDAPTIPVISTVTDSVAPVTGAITAGGSTNDAMPVLTGTAEANSTISVFDGTTLLGTTTADASGNWTFTPTTALTDGSHSLTATATDPAGNVSTTSSAFALTVDTTAPAAPVITTVTDAVSPVTGAITAGGSTNDAAPALTGTAEAYSTISVFDGTTLLGTTTADASGNWTFTPTT
ALTDGSHSLSATATDATGNVSTASSAFTLTVDTDAPATPVISTVTDDVAPVTGAITAGGSTNDATPTLAGTAEANSTVSIFDGTTLLGTTTADASGSWTFTPTTALTDGSHSLTATVTDAAGNVSTASTAFTLTVDTAAPAAPVLNPTTGTTLSGTAEANSTISVSDGTTLLGTTTADASGTWTYTPTTP
LASGTVVSATATDPAGNVSPAATVTVTAVNTAPAAPVISTVTDDVAPITGAITAGGSTNDATPTLTGTAEADSTISVFDGTTLLGTTTADASGNWTFTPTTALTDGSHSLTATATNATGDVSPASSAFTLTVATAAPATPVITTVTDDVAPVTGAIAAAGSTNDATPTLAGTAVANSTVSVFDGTTLLGT
TTADASGNWTFTPTTALTDGSHSFTATATDAAGNVSAASSAFTLTVDTAAPATPVVGTVTDSVAPVTGAITAGGTTNDATPTLTGTAEANSTISIFDGITLVGTTTADASGNWTFSPTTALADGSHSLTATATDAAGNVSTASSAFALTVDTAAPAAPVLVSVTDNVAPLTGTIAAGGATNDATPTLTGT
AEANSTISVFDGTTLLGTTTADASGNWTFTPTTSLADGSRSLTATTTDTAGNVSTASTAFTLTVDTDAPPTPVITTVTDNVAPVTGAVTAGGSTNDATPTLTGTAEAHNMISIFDGATLIGATVADASGNWTFTPIQPLADGNHSLTATATDPAGNVGTASSTFTLTVDTAAPAAPVISTVTDNVAPVTG
AITAGGSTNDAMPVLTGTAEANSTVSILDGTTLLGTTTADASGNWTFTPTTALTDGSHSITATATDAAGNVSTASSAFAMTVDTAAPATPVITTVTDDVAPVTGAITAGGSTNDATPTLTGTAEANSTISIFDGSTLLGTVTADASGNWTYTPTTALADGSHSFTATATDAAGNVSTATSAFTLTVDTAA
PATPALVSVTDDVAPVTGVLTSGGSTNDATPTLTGTAEANSTVSILDGTTLLGTTTADASGNWTFTPTTPLADGSHSLTATATDAAGNVSTATSAFTLTVDTAAPATPVLASVTDDVAPVTGALTSGGSTNDATPTLAGTAEANSTISILDGTTLLGTTTADASGNWTFTPTTALADGSHSLTATATDAA
GNVSTATSAFTLTVDTAAPATPVLVSVTDDVAPVTGVLTSGGSTNDATPTLTGTAEANSTISVFDGTTLLGTTTADASGNWTFTPTTGLTDGSHSLTATATDAAGNVSPATSAFTQTVDTAAPATPALVSVTDDVAPVTGVLTSGGSTNDATPTLAGTAEANSTISILDGTTLLGTTTADASGSWTFTPT
TGLTDGSHSLTATATDAAGNVSTATTAFTLTVDTVAPTTTATLTTLTADTGTTGDWITGDTAPTLSGTVSAALGTGETVQVSLDGGTTWSDAVVSGTSWSWYPVAQFAAGGYTAMTRVVDAAGNAGAASSQAFTIQTTAAPVVSVQQSAGLLGIAGANLFDLIDFSQQQLFAATDYNNNIQSVVLHYGGV
ASLLSTSQYSANSALASELGLQFSVVNNNGILGIAASSTMTITATDGGTIDNLRLNEFLGSVTLSGGGLLGINASLLSSVTIQATDSTSLSSTANAGQLLNVSLLGTTQPSAIIEGTSGNNTLTGTSGNDRLYGYAGDDTLNAGAGNDLLRGGAGNDVLNGGDGNDILIGGAGNDTLTGGAGGDIFLWEV
NAADNTGGNGADTITDFTLASGPDDATADRIDVSALLVGYTADADGAAHYVNGVATIDSGDTIGQYLTATSDGAGNTVITIDRDGAGTTYSATTLVTLTNVTTNLETLLANHQILV
Molecular weight: 395.09 kDa
Isoelectric point according different methods:
Isoelectric point according different methods:
Protein with the highest isoelectric point:
>tr|Q8XZ87|Q8XZ87_RALSO Uncharacterized protein
MVLMLPQARSGAQVRAVRVSAMSRPVASAVRVPVAKVAGKPVARAKPVASASAKRRKR
MVLMLPQARSGAQVRAVRVSAMSRPVASAVRVPVAKVAGKPVARAKPVASASAKRRKR
Molecular weight: 6.08 kDa
Isoelectric point according different methods:
Isoelectric point according different methods:
General Statistics
Number of major isoforms |
Number of additional isoforms |
Number of all proteins |
Number of amino acids |
Min. Seq. Length |
Max. Seq. Length |
Avg. Seq. Length |
Avg. Mol. Weight |
|---|---|---|---|---|---|---|---|
1,654,857 |
20 |
6,889 |
332.7 |
36.0 kDa |
Amino acid frequency
Ala |
Cys |
Asp |
Glu |
Phe |
Gly |
His |
Ile |
Lys |
Leu |
Met |
Asn |
Gln |
Pro |
Arg |
Ser |
Thr |
Val |
Trp |
Tyr |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
13.43 |
0.94 |
5.4 |
4.89 |
3.37 |
8.42 |
2.32 |
4.36 |
2.83 |
10.31 |
2.33 |
2.69 |
3.89 |
5.39 |
7.27 |
5.32 |
5.57 |
7.5 |
1.41 |
2.36 |
For dipeptide frequency statistics click here
Proteome-pI is available under Creative Commons Attribution-NoDerivs license, for more details see here
| Reference: Kozlowski LP. Proteome-pI: proteome isoelectric point database. Nucleic Acids Res. 2016. doi: 10.1093/nar/gkw978 |
Contact: Lukasz P. Kozlowski |

