Kazachstania africana (strain ATCC 22294 / BCRC 22015 / CBS 2517 / CECT 1963 / NBRC 1671 / NRRL Y-8276) (Yeast) (Kluyveromyces africanus)
Taxonomy: cellular organisms; Eukaryota; Opisthokonta; Fungi; Dikarya; Ascomycota; saccharomyceta; Saccharomycotina; Saccharomycetes; Saccharomycetales; Saccharomycetaceae; Kazachstania; Kazachstania africana
Average proteome isoelectric point is 6.51
Get precalculated fractions of proteins
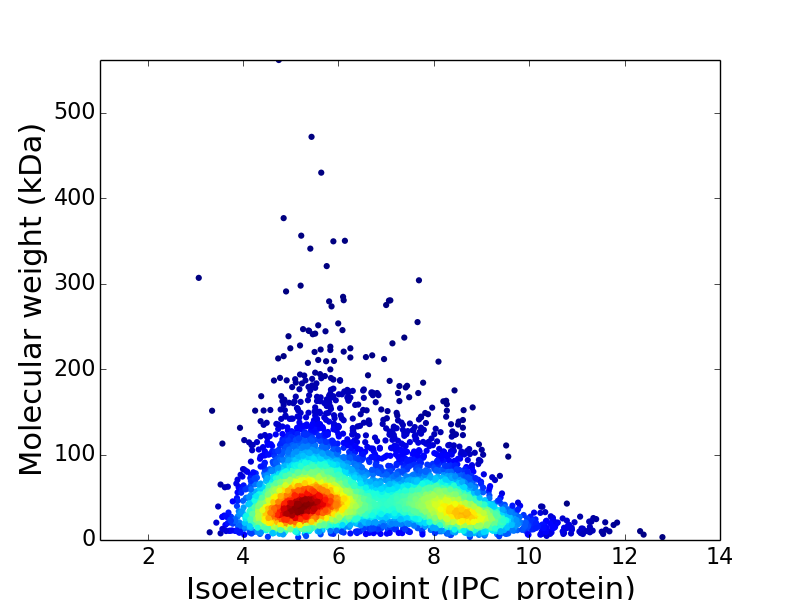
Virtual 2D-PAGE plot for 5,354 proteins (isoelectric point calculated using IPC_method)
Get csv file with sequences according given criteria:
* You can choose from 18 different methods for calculating isoelectric point
Protein with the lowest isoelectric point:
>tr|H2B2H5|H2B2H5_KAZAF Uncharacterized protein (Fragment)
MRLIKLLTIFIQISTYFVNALDVSSCANIYGSTFFHESVTVRPNKGLTLETGLWHFFWRAVINEGDLFIRETNNLLLGTSINFFSGTSNEGNIVVDDTTATTAGTMSFSDTTHNFGNIWFAGKNTLLTSVSFSMTGKIIENTGLIYVKQTNTGNSGGSFTMGKSSSTVTNDGTLCAEEVTLSPGATIYGS
GCITLLSGSTLNIDNIKSYPLSGQTIYMNSTSALIYITHKSTTASLTITGFGKSNKIQLSQSIDSYSYDSTTGILTVVSKVFLLGTLTLNLNIGTGYASSGFSTSASGLLTGKDTITYSPAAPATSLPDGCSACLAAPTERNFCSGSSVSSMSNTFTDDSEESTTYEISYSSSDTLSYTVSTSDYSSSFS
SITNPSKTTEISSTPSSKSASESPSETSSLSSANYYTTTNSLDATATGSYSGTGSLDATATGSYSGTDSLDATATGSYSGTNSLDATATGSYSGTNSLDATATGSYSGTDSVDATATGSYSGTNSLDATATGSLSGTDSVDATATGSYSRTNSVDPTATGSYSGTNSLDATATGSLSGTNSLDATATGSY
SGTNSLDATATGSLSGTDSVDATATGSYSRTNSLDATATGSYSGTNSLDATATGSYSGTNSLDATATGSYSGTNSLDATATGSYSGSNSVDPTATGSYSGTDSLDATATGSLSGTNSLDATATGSLSGSNSVDPTATGSYSGTNSLDATATGSYSGSNSLDATVTGSLSGTNSVDATATGSYSGTNSLDA
TATGSYSRTNSLDATATGSYSGTNSLDATATGSLSGSNSVDPTATGSYSGTDSLDATATGSYSGTNSVDGTATDSQSGTVILTPSSASSYSGTNSLDATATGSYSGTNSLDATATGSYSGTNSLDATATGSLSGSNSVDPTATGSYSGTNSLDATATGSLSGTNSLDATATGSYSGTNSLDATATGSYSG
TNSLDATATGSYSGTNSLDATATGSYSGSNSLDATFTGSYSGTNSVDATATGSYSGTNSVDATATGSYSGTNSLDATATGSYSGTNSVDATATGSLSGTNSLEATATGSYSGSNSLDATATGSYSGTNSIDSTATNSQSGTVILTPSSTNSYSGSNSLDATATGSYSGSNSLDATFTGSYSGSNSLDATA
TGSYSGTNSIDSTATNSQSGTVILTPSSTNSYSGSNSLDATATGSYSGSNSLDATATGSLSGTNSVDATATGSYSGTNSVDATATGSYSGSNSVDATATGSYSGTNSVDATATGSYSGSNSLDATATGSYSGTNSIDSTATNSQSGTVILTPSSTNSYSGSNSLDATATGSYSGSNSLDATFTGSYSGSN
SLDATATGSYSGTNSVDATATGSYSGSNSLDATFTGSYSGTNSVDATATGSYSGTNSVDATATGSLSGTNSLDATATGSYSGSNSVDPTATGSYSGTNSLDATATGSLSGSNSVDPTATGSYSGTNSLDATATGSLSGTNSLDATATGSYSSSNSLDATFTGSYSGSNSLDATATGSYSGSNSLDATATG
SYSGSNSLDATATGSFSGSNSIDSTATNSQSGTVILTPSSTNSYSGSNSLDATATGSYSGTNSLDATATGSYSGSNSLDATFTGSYSGTNSVDATATGSYSGTNSVDATFTGSYSGSNSLDATATGSYSGSNSLDATATGSLSGSNSVDPTATGSYSGTDSLDATATGSLSGTNSLEATATGSLSGSNSV
DPTATGSYSGTDSLDATATDSLSGTDSVDATATGSYSGTNSLDATATGSLSGTNSLDATATGSLSGSNSVDATATGSYSGTDSLDVSATGSLSGSNSVDPTATGSYSGTNSLDATFTGSYSGSNSLDATATGSYSGSNSLDATATGSLSGSNSVDPTATGSYSGTDSLDATATGSLSGTDSVDATATGSY
SGTNSLDATATGSLSGTNSLDATATGSLSGSNSVDPTATGSYSGTNSLDATATGSLSGTNSLDATATGSYSGSNSVDPTATGSYSGTNSLDATATGSLSGSNSVDGTATDSQSGTVILTPSSASSYSGTNSLDATATGSLSGSNSVDPTATGSYSGTNSLDATATGSLSGTDSVDATATGSYSRTNSLDA
TATGSLSGSNSVDPTATGSYSGTNSLDATATGSYSGTNSLDATATGSYSGTDSLDATATGSLSGSNSVDPTATGSYSRTNSLDATATGSLSGSNSVDPTATGSYSGTNSVDGTATDSQSGTVILTPSSASSYSGTNSLDATATGSLSGTNSLDATATGSYSGTDSVDATATGSYSGTNSPDATATGSYSG
TNSLDATATGSYSGTDSLDVSATGSLSGSNSVDPTATGSYSGTNSLDATATGSYSGSNSVDPTATGSYSGTNSLDATATGSLSGSNSLDATATGSYSGTNSIDSTATNSQSGTVILTPSSTNSYSGSNSLDATATGSYSGTNSLDATATGSYSGSNSLDATFTGSYSGTNSVDATATGSYSGTNSVDATF
TGSYSGSNSLDATATGSYSGSNSLDATATGSLSGSNSVDPTATGSYSGTDSLDATATGSLSGTNSLDATATGSLSGSNSVDPTATGSYSGTNSLDATATGSLSGTNSLDATATGSYSGPNSVDPTATGSYSGTNSLDATFTGSYSGSNSLDATATGSYSGSNSLDATATGSYSGTNSIDSTATNSQSGTV
ILTPSSTNSYSGSNSLDATATGSYSGSNSLDATFTGSYSGSNSLDATFTGSYSGSNSLDATATGSYSGTNSIDSTATNSQSGTVILTPSSTNSYSGSNSLDATVHLDHTLVPTLVDATATGSYSGSNSIDSTATNSQSGTVILTPSSTNSYSGSNSLDATATGSYSGSNSLDATATGSYSGSNSLDATAT
GSYSGSNSLDATATGSYSGTNSLDATATGSLSGTNSLDATATGSLSGSNSVDPTATGSYSGTNSLDATATGSLSGTNSLDATATGSYSGSNSVDPTATGSYSGTDSLDATATGSLSGTDSVDATATGSYSGTNSLDATATGSLSGTNSLDATATGSLSGSNSVDPTATGSYSGTDSLDATATGSLSGTNS
LDATATGSYSSSNSLDATFTGSYSGSNSLDATATGSLSGSNSVDGTATDSQSGTVILTPSSASSYSGTNSLDATATGSLSGSNSVDPTATGSYSGTNSLDATATGSLSGTDSVDATATGSYSRTNSLDATATGSLSGSNSVDPTATGSYSGTNSLD
MRLIKLLTIFIQISTYFVNALDVSSCANIYGSTFFHESVTVRPNKGLTLETGLWHFFWRAVINEGDLFIRETNNLLLGTSINFFSGTSNEGNIVVDDTTATTAGTMSFSDTTHNFGNIWFAGKNTLLTSVSFSMTGKIIENTGLIYVKQTNTGNSGGSFTMGKSSSTVTNDGTLCAEEVTLSPGATIYGS
GCITLLSGSTLNIDNIKSYPLSGQTIYMNSTSALIYITHKSTTASLTITGFGKSNKIQLSQSIDSYSYDSTTGILTVVSKVFLLGTLTLNLNIGTGYASSGFSTSASGLLTGKDTITYSPAAPATSLPDGCSACLAAPTERNFCSGSSVSSMSNTFTDDSEESTTYEISYSSSDTLSYTVSTSDYSSSFS
SITNPSKTTEISSTPSSKSASESPSETSSLSSANYYTTTNSLDATATGSYSGTGSLDATATGSYSGTDSLDATATGSYSGTNSLDATATGSYSGTNSLDATATGSYSGTDSVDATATGSYSGTNSLDATATGSLSGTDSVDATATGSYSRTNSVDPTATGSYSGTNSLDATATGSLSGTNSLDATATGSY
SGTNSLDATATGSLSGTDSVDATATGSYSRTNSLDATATGSYSGTNSLDATATGSYSGTNSLDATATGSYSGTNSLDATATGSYSGSNSVDPTATGSYSGTDSLDATATGSLSGTNSLDATATGSLSGSNSVDPTATGSYSGTNSLDATATGSYSGSNSLDATVTGSLSGTNSVDATATGSYSGTNSLDA
TATGSYSRTNSLDATATGSYSGTNSLDATATGSLSGSNSVDPTATGSYSGTDSLDATATGSYSGTNSVDGTATDSQSGTVILTPSSASSYSGTNSLDATATGSYSGTNSLDATATGSYSGTNSLDATATGSLSGSNSVDPTATGSYSGTNSLDATATGSLSGTNSLDATATGSYSGTNSLDATATGSYSG
TNSLDATATGSYSGTNSLDATATGSYSGSNSLDATFTGSYSGTNSVDATATGSYSGTNSVDATATGSYSGTNSLDATATGSYSGTNSVDATATGSLSGTNSLEATATGSYSGSNSLDATATGSYSGTNSIDSTATNSQSGTVILTPSSTNSYSGSNSLDATATGSYSGSNSLDATFTGSYSGSNSLDATA
TGSYSGTNSIDSTATNSQSGTVILTPSSTNSYSGSNSLDATATGSYSGSNSLDATATGSLSGTNSVDATATGSYSGTNSVDATATGSYSGSNSVDATATGSYSGTNSVDATATGSYSGSNSLDATATGSYSGTNSIDSTATNSQSGTVILTPSSTNSYSGSNSLDATATGSYSGSNSLDATFTGSYSGSN
SLDATATGSYSGTNSVDATATGSYSGSNSLDATFTGSYSGTNSVDATATGSYSGTNSVDATATGSLSGTNSLDATATGSYSGSNSVDPTATGSYSGTNSLDATATGSLSGSNSVDPTATGSYSGTNSLDATATGSLSGTNSLDATATGSYSSSNSLDATFTGSYSGSNSLDATATGSYSGSNSLDATATG
SYSGSNSLDATATGSFSGSNSIDSTATNSQSGTVILTPSSTNSYSGSNSLDATATGSYSGTNSLDATATGSYSGSNSLDATFTGSYSGTNSVDATATGSYSGTNSVDATFTGSYSGSNSLDATATGSYSGSNSLDATATGSLSGSNSVDPTATGSYSGTDSLDATATGSLSGTNSLEATATGSLSGSNSV
DPTATGSYSGTDSLDATATDSLSGTDSVDATATGSYSGTNSLDATATGSLSGTNSLDATATGSLSGSNSVDATATGSYSGTDSLDVSATGSLSGSNSVDPTATGSYSGTNSLDATFTGSYSGSNSLDATATGSYSGSNSLDATATGSLSGSNSVDPTATGSYSGTDSLDATATGSLSGTDSVDATATGSY
SGTNSLDATATGSLSGTNSLDATATGSLSGSNSVDPTATGSYSGTNSLDATATGSLSGTNSLDATATGSYSGSNSVDPTATGSYSGTNSLDATATGSLSGSNSVDGTATDSQSGTVILTPSSASSYSGTNSLDATATGSLSGSNSVDPTATGSYSGTNSLDATATGSLSGTDSVDATATGSYSRTNSLDA
TATGSLSGSNSVDPTATGSYSGTNSLDATATGSYSGTNSLDATATGSYSGTDSLDATATGSLSGSNSVDPTATGSYSRTNSLDATATGSLSGSNSVDPTATGSYSGTNSVDGTATDSQSGTVILTPSSASSYSGTNSLDATATGSLSGTNSLDATATGSYSGTDSVDATATGSYSGTNSPDATATGSYSG
TNSLDATATGSYSGTDSLDVSATGSLSGSNSVDPTATGSYSGTNSLDATATGSYSGSNSVDPTATGSYSGTNSLDATATGSLSGSNSLDATATGSYSGTNSIDSTATNSQSGTVILTPSSTNSYSGSNSLDATATGSYSGTNSLDATATGSYSGSNSLDATFTGSYSGTNSVDATATGSYSGTNSVDATF
TGSYSGSNSLDATATGSYSGSNSLDATATGSLSGSNSVDPTATGSYSGTDSLDATATGSLSGTNSLDATATGSLSGSNSVDPTATGSYSGTNSLDATATGSLSGTNSLDATATGSYSGPNSVDPTATGSYSGTNSLDATFTGSYSGSNSLDATATGSYSGSNSLDATATGSYSGTNSIDSTATNSQSGTV
ILTPSSTNSYSGSNSLDATATGSYSGSNSLDATFTGSYSGSNSLDATFTGSYSGSNSLDATATGSYSGTNSIDSTATNSQSGTVILTPSSTNSYSGSNSLDATVHLDHTLVPTLVDATATGSYSGSNSIDSTATNSQSGTVILTPSSTNSYSGSNSLDATATGSYSGSNSLDATATGSYSGSNSLDATAT
GSYSGSNSLDATATGSYSGTNSLDATATGSLSGTNSLDATATGSLSGSNSVDPTATGSYSGTNSLDATATGSLSGTNSLDATATGSYSGSNSVDPTATGSYSGTDSLDATATGSLSGTDSVDATATGSYSGTNSLDATATGSLSGTNSLDATATGSLSGSNSVDPTATGSYSGTDSLDATATGSLSGTNS
LDATATGSYSSSNSLDATFTGSYSGSNSLDATATGSLSGSNSVDGTATDSQSGTVILTPSSASSYSGTNSLDATATGSLSGSNSVDPTATGSYSGTNSLDATATGSLSGTDSVDATATGSYSRTNSLDATATGSLSGSNSVDPTATGSYSGTNSLD
Molecular weight: 306.91 kDa
Isoelectric point according different methods:
Isoelectric point according different methods:
Protein with the highest isoelectric point:
>tr|H2B284|H2B284_KAZAF Uncharacterized protein
MRAKWRKKRVRRLKRKRRKVRARSK
MRAKWRKKRVRRLKRKRRKVRARSK
Molecular weight: 3.34 kDa
Isoelectric point according different methods:
Isoelectric point according different methods:
General Statistics
Number of major isoforms |
Number of additional isoforms |
Number of all proteins |
Number of amino acids |
Min. Seq. Length |
Max. Seq. Length |
Avg. Seq. Length |
Avg. Mol. Weight |
|---|---|---|---|---|---|---|---|
0 |
2,609,361 |
25 |
4,933 |
487.4 |
55.3 kDa |
Amino acid frequency
Ala |
Cys |
Asp |
Glu |
Phe |
Gly |
His |
Ile |
Lys |
Leu |
Met |
Asn |
Gln |
Pro |
Arg |
Ser |
Thr |
Val |
Trp |
Tyr |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
5.19 |
1.18 |
5.99 |
6.7 |
4.55 |
4.7 |
2.04 |
6.99 |
7.41 |
9.72 |
2.06 |
6.58 |
3.76 |
3.99 |
4.27 |
8.91 |
5.95 |
5.52 |
1.01 |
3.46 |
For dipeptide frequency statistics click here
Proteome-pI is available under Creative Commons Attribution-NoDerivs license, for more details see here
| Reference: Kozlowski LP. Proteome-pI: proteome isoelectric point database. Nucleic Acids Res. 2016. doi: 10.1093/nar/gkw978 |
Contact: Lukasz P. Kozlowski |

